计算智能导论作业——遗传算法的实现
目 录
1 背景知识 1
1.1 最优化问题 1
1.2 进化算法 1
1.3 遗传算法的基本知识 1
1.3.1 生物背景 2
1.3.2 基本思想 2
1.4 遗传算法的组成部分 2
1.4.1 编码机制 3
1.4.2 种群初始化 3
1.4.3 适应度函数 3
1.4.4 遗传算子 3
2 算法步骤 4
3 实验过程 4
3.1 代码实现 4
4 结果分析 5
5 总结 5
A 程序代码 7
1 背景知识
1.1 最优化问题
工程设计中最优化问题 (optimization problem) 的一般提法是要选择一组参数 (变量),
在满足一系列有关的限制条件 (约束) 下, 使设计指标 (目标) 达到最优值。
最优化问题一般包括两方面问题: 线性问题和非线性问题。一方面是线性问题的求解, 主要在经济活动及工程技术中出现。这类问题一般采用单纯形法来求解。另一方面是非线性问题的求解, 这类问题在工程中经常碰到, 是最为常见的一类问题, 尤其是在物理学和决策中, 许多问题常常可以归结为非线性规划问题。这类问题一般需要先建立一个数学模型, 再进行求解。
最优化问题的求解实质就是将物理问题数学化, 把最优化问题的求解转化为目标函数
最优解的求解, 利用遗传算法在求解最优解方面的特点, 达到事半功倍的效果。
1.2 进化算法
进化算法, 或 “演化算法” (evolutionary algorithms) 是一个 “算法簇”, 是在达尔文 (Darwin) 的进化论和孟德尔 (Mendel) 的遗传变异理论的基础上产生的一种在基因和种群层次上模拟自然界生物进化过程与机制, 进行问题求解的自组织、自适应的随机搜索技术。它以达尔文进化论的 “物竞天择、适者生存” 作为算法的进化规则, 并结合孟德尔的遗传变异理论, 将生物进化过程中的繁殖 (Reproduction), 变异 (Mutation), 竞争 (Competition)、选择 (Selection) 引入到了算法中, 是一种对人类智能的演化模拟, 主要有遗传算法、演化策略、演化规划和遗传规划四大分支。
尽管它有很多的变化, 有不同的遗传基因表达方式, 不同的交叉和变异算子, 特殊算子的引用, 以及不同的再生和选择方法, 但它们产生的灵感都来自于大自然的生物进化。
与传统的基于微积分的方法和穷举法等优化算法相比, 进化计算是一种成熟的具有高鲁棒性和广泛适用性的全局优化方法, 具有自组织、自适应、自学习的特性, 能够不受问题性质的限制, 有效地处理传统优化算法难以解决的复杂问题。
1.3 遗传算法的基本知识
遗传算法 (Genetic Algorithm, GA) 是进化算法的一个分支, 是一种模拟自然界生物进化过程的随机搜索算法。其将 “优胜劣汰, 适者生存” 的生物进化原理引入优化参数形成的编码串联群体中, 按所选择的适应度函数并通过遗传中的复制、交叉及变异对个体进行筛选, 使适应度高的个体被保留下来, 组成新的群体。
新的群体既继承了上一代的信息, 又优于上一代, 这样周而复始, 群体中个体适应度不断提高, 直到满足一定的条件。同时, 遗传算法的原理简单, 可并行处理, 并能得到全局最优解。
遗传算法是模拟达尔文生物进化论的自然选择和遗传学机理的生物进化过程的计算模型, 是一种通过模拟自然进化过程搜索最优解的方法。该算法通过数学的方式, 利用计算机仿真运算, 将问题的求解过程转换成类似生物进化中的染色体基因的交叉、变异等过程。在求解较为复杂的组合优化问题时, 相对一些常规的优化算法, 通常能够较快地获得较好的优化结果。遗传算法已被人们广泛地应用于组合优化、机器学习、信号处理、自适应控制和人工生命等领域。
遗传算法直接以目标函数值作为搜索信息。传统的优化算法往往不只需要目标函数值,
还需要目标函数的导数等其它信息。这样对许多目标函数无法求导或很难求导的函数, 遗传算法就比较方便。
同常规算法相比, 遗传算法有以下特点:
遗传算法是对决策变量的编码进行操作, 这样提供的参数信息量大, 优化效果好。遗传算法是从许多点开始并行操作, 因而可以有效地防止搜索过程收玫于局部最优解。遗传算法通过目标函数来计算适配值, 而不需要其他推导和附加信息, 从而对问题的依赖性小。遗传算法的寻优规则是由概率决定的, 而非确定性的。遗传算法在解空间进行高效启发式搜索, 而非盲目地穷举或完全随机搜索。遗传算法对于待寻优的函数基本无限制, 因而应用范围较广。遗传算法具有并行计算的特点, 因而可通过大规模并行计算来提高计算速度。遗传算法更适合大规模复杂问题的优化。遗传算法计算简单, 功能强。
1.3.1 生物背景
- 基因: 一个遗传因子。
- 染色体: 包含一组的基因。
- 个体: 组成种群的单个生物。
- 种群: 生物的进化以群体的形式进行, 这样的一个群体称为种群。
- 生存竞争, 适者生存: 对环境适应度高的个体参与繁殖的机会比较多, 后代就会越来越多: 适应度低的个体参与繁殖的机会比较少, 后代就会越来越少。
1.3.2 基本思想
- 遗传算法把问题的解表示成 “染色体”, 在算法中即是以一定方式编码的串。
- 在执行遗传算法之前, 给出一群 “染色体”, 也即一组候选解 (种群)。
- 把这些假设解置于问题的 “环境” 中, 并按适者生存的原则, 从中选择出较适应环境的 “染色体” 进行复制, 再通过交叉, 变异过程产生更适应环境的新一代 “染色体”群。
这样, 一代一代地进化, 最后就会收玫到最适应环境的一个 “染色体” 上, 它就是求解
到的最优解。
1.4 遗传算法的组成部分
一般的遗传算法由四个部分组成: 1. 编码机制 2. 种群初始化 3. 适应度函数
4. 遗传算子 (选择、交叉、变异)
1.4.1 编码机制
用遗传算法解决问题时, 首先要对待解决问题的模型结构和参数进行编码, 一般用字符串表示。GA 中的编码方法: 二进制编码、浮点数编码方法、格雷码编码、符号编码等。
二进制编码所构成的个体的基因型是一个由 0 或 1 组成的编码串,其符号串的长度 $L$ 与问题所要求的求解精度有关。设某一变量的取值范围是 $\left[ X_{\min}, X_{\max} \right]$, $X_{\min} < X_{\max}$ 则二进制编码的编码精度为:
$$
\delta = \frac{X_{\max} - X_{\min}}{2^{L} - 1}
$$
如果某一个体的编码是: $b_{L}b_{L - 1}b_{L - 2} \cdots b_{2}b_{1}$, 则对应的解码公式为:
$$
X = X_{\min} + \frac{X_{\max} - X_{\min}}{2^{L} - 1} \left( \sum_{i = 1}^L b_{i} 2^{i - 1} \right)
$$
1.4.2 种群初始化
对种群个体的编码进行随机初始化。首先, 我们需要建立种群个体与遗传算法中有效解的对应概念。其次针对于每个个体,在固定长度的染色体下,对每个基因位进行随机 (0/1) 赋值, 也就是对有效解进行初始化的过程。
1.4.3 适应度函数
遗传算法以个体适应度的大小来评定各个个体的优劣程度, 从而决定其遗传机会的大小。一般情况下, 可以将目标函数作为适应度函数, 也就是求解问题的优化目标。将种群中的个体的染色体进行解码, 可以得到对应的十进制值, 代入适应度函数即可求得适应值。
1.4.4 遗传算子
(1)选择操作
选择是对当前种群内的所有个体进行筛选, 流出进入到后续繁殖环节的父代个体的过程, 常采用轮盘赌选择。轮盘赌选择依据个体的适应度值计算每个个体在下一代中出现的概率, 并按照此概率随机选择个体构成子代种群。选择某个体的概率为
$$
p(I_{i}) = \frac{f(I_{i})}{\sum_{i = 1}^N f(I_{i})}
$$
其中, $f(I_{i})$ 是个体 $I_{i}$ 的适应度值, $N$ 是种群大小。轮盘赌选择的流程如下:
- 计算种群中所有个体的适应度值之和, $F = \sum_{k = 1}^N \text{eval}(v_{k})$ ;
- 计算每个个体 $v_{k}$ 的选择概率 $p_{k}$, $p_{k} = \frac{\text{eval}(v_{k})}{F}$, $k = 1, 2, \ldots, \text{popSize}$ ;
- 计算每个个体 $v_{k}$ 的累积概率 $q_{k}$, $q_{k} = \sum_{j = 1}^k p_{j}$, $k = 1, 2, \ldots, \text{popSize}$ ;
- 随机产生 $N$ 个 $[0,1]$ 的随机数 $r_{i}$ ;
- 对于每一个 $r_{i}$ : 如果 $r_{i} \leq q_{1}$, 选择第一个个体 $v_{1}$; 否则, 如果 $q_{k - 1} < r_{i} < q_{k}$, 选择第 $k$ 个个体 $v_{k}(2 \leq k \leq \text{popSize})$ 。
(2)交叉操作
交叉操作是选中的两个父代个体交换莱些基因位形成子代个体的过程。交叉概率 (P_{c}) 是在种群中个体被选择出进行交叉的概率, 一般的交叉方式有单点交叉、两点交叉、多点交叉、均匀交叉等。其中, 单点交叉是随机产生一个有效的交叉位置, 染色体交换位于该交叉位置后的所有基因。
(3)变异操作
变异操作是编码按小概率扰动产生的变化,类似于基因的突变。变异概率 (P_{m}) 是控制算法中变异操作的使用频率。常用的变异方式有单点变异、均匀变异、高斯变异等。其中, 单点变异指的是对于某一基因位,产生的随机数小于 (P_{m}) 则改变该基因的取值,否则该基因不发生变异, 保持不变。
2 算法步骤
该算法可分为以下 7 步。
Step1: 初始化种群;
Step2: 计算种群中每个个体的适应度值;
Step3: 按由个体适应度值决定的某个规则选择将进入下一代的个体; Step4: 按概率 (P_{c}) 进行交叉操作; Step5: 按概率 (P_{m}) 进行变异操作;
Step6: 若没有满足某种终止条件, 则转第 2 步, 否则进入下一步; Step7: 输出种群中适应度值最优的染色体作为问题的满意解或最优解。
3 实验过程
本文中采用的函数为 (f(x) = {\sum}{i = 1}^n\left\lbrack x{i}^{2} {-} 10\cos\left( 2\pi x_{i} + 10 \right) \right\rbrack) ,自变量的定义域为 (\lbrack{-}5.12,5.12\rbrack) , 函数的最小值为 0 。
3.1 代码实现
作为遗传算法中较为重要的交叉模块, 我们进行了如下设计。首先, 遍历种群中的每一个个体, 将该个体作为父代, 而子代首先得到父代的全部基因, 父代产生子代时不是必然发生交叉, 而是以一定的概率发生交叉。从种群中选择令一个个体, 将该个体作为另一个附带, 随机产生交叉点后, 子代得到此父代交叉点后的全部基因。同时, 此子代也具有相应的变异概率。
这里首先确定了相关参数, 然后定义主要的几个函数, 包括三维作图函数、解码函数、适应度计算函数、交叉变异函数, 然后进行操作整合, 并将函数整合起来, 代码见附录。
在本代码中, 不仅对遗传算法中不同个体的适应度情况实时展示, 而且对迭代过程中
最小的值进行记录, 画出折线图, 从而使得展示过程更加直观。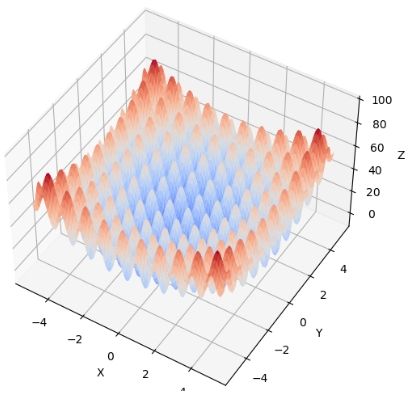
图 1: 函数图
4 结果分析
上图为运算过程中的函数以及整个种群的情况:
当迭代完成后, 可以看到仅函数中心处有一点, 说明整个种群都收玫到了一处, 如下图
所示。
下图为迭代过程的最小适应度函数值的变化折线图。
由图中可以看出, 所求适应度函数最小的值在不断下降, 最终达到稳定值, 而且最终所
处数值接近 5 , 与题中一直条件相吻合, 证明了该遗传算法解决问题的效果较好。
最终得到最优的基因型为 [1100000000000000000001100100101111110100010100001], 其中 (x) 和 (y) 分别为 0.006485290913897046,0.003113098330086217,此时的值为: 0.01027 。
5 总结
在这次实验中, 开始在理解题意方面遇到了很多问题, 后来经过多方询问才明白。这次实验中我通过广泛查询资料了解到了相关的知识, 也认真写代码来完成任务, 这份作业的完成确实比较艰巨, 一份顶多份, 但是我还是有很大的收获, 能力也得到了提升。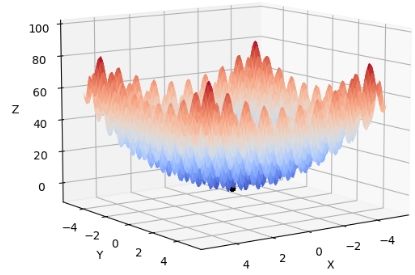
图 2: 函数图
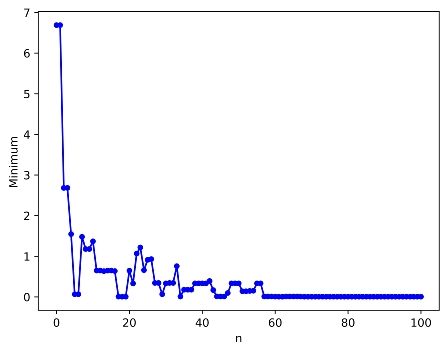
图 3: 函数图
A 程序代码
遗传算法程序 - GA.py
1 | import math |





