数据挖掘报告——MIMC数据集的预处理
摘 要
在本次的大作业中,这里首先利用 MySQL 对数据进行整合,并提取出包含 $CO_2$ 和 $O_2$ 相应指标的数据,然后将提取的数据导出,并导入到 Python 中。然后利用第一组数据为示例分别进行缺失值、离群点处理,并完成去噪以及插值处理。随后选出另两组为示例进行可视化,最后得出实验感想与分析,这里去噪利用了 $3\sigma$ 原则。
关键词: $\quad$ MySQL Python $\quad 3\sigma$ 原则
目 录
1 数据处理 1
1.1 数据提取 1
1.2 数据具体处理 1
1.3 数据合并 1
1.4 缺失值处理 2
1.5 离群点处理 2
1.6 去噪 4
1.7 插值 5
2 第二组数据 5
2.1 离群点处理 5
2.2 去噪 6
2.3 插值 7
3 第三组数据 8
3.1 离群点处理 8
3.2 去噪 9
3.3 插值 9
4 总结 10
A SQL 程序代码 1 11
B SQL 程序代码 2 11
(\mathrm{C}) 主程序代码 11
D 作业要求 14
D. 1 数据集说明 14
D. 2 任务说明 14
1 数据处理
1.1 数据提取
这次作业中数据较大, 直接读取很难看出数据具体细节, 直接打开会崩溃, 因此这里开始使用 MySQL, 将提供的三个文件导入进去, 然后进行初步处理, 利用附录 A 和附录 B 中所示代码, 将 PO2 和 PCO2 两个指标提取出来, 得到数据 CO2 如下图所示。
| f1 | ROW ID | SUBJECT ID | HADM ID | ITEMID | CHARTTIMEVALUE | VALUENUM | VALUEUOM | FLAG | time |
|---|---|---|---|---|---|---|---|---|---|
| 974 | 3 | 145834.0 | 50818 | 2101-10-2204:31:0025 | 25.0 | mmHg | (Null) | 0 | |
| 231 | 988 | 3 | 145834.0 | 50818 | 2101-10-2207:13:0028 | 28.0 | mmHg | (Null) | 0 |
| 240 | 997 | 3 | 145834.0 | 50818 | 2101-10-2210:16:0028 | 28.0 | mmHg | (NuII) | 0 |
| 249 | 1006 | 3 | 145834.0 | 50818 | 2101-10-2211:21:0027 | 27.0 | mmHg | (Null) | 0 |
| 262 | 1019 | 3 | 145834.0 | 50818 | 2101-10-2213:02:0026 | 26.0 | mmHg | CNUII | 0 |
| 271 | 1028 | a | 145B34.0 | 50818 | 2101-10-2215:59:0024 | 24.0 | mmHg | (NuII) | 0 |
| 285 | 1042 | 3 | 145834.0 | 50818 | 2101-10-2216:02:0030 | 30.0 | mmHg | (Nuith | 0 |
| 310 | 1067 | 3 | 145834.0 | 50818 | 2101-10-2221:20:0027 | 27.0 | mmHg | (Null) | 0 |
| 325 | 1082 | 3 | 145834.0 | 50818 | 2101-10-2302:46:0029 | 29.0 | mmHg | (Null) | 0 |
| 338 | 1095 | 3 | 145834.0 | 50818 | 2101-10-2302:49:0033 | 33.0 | mmHg | (Null) | 0 |
| 379 | 1136 | 3 | 145834.0 | 50818 | 2101-10-2310:14:0025 | 25.0 | mmHg | (Null) | 0 |
| 392 | 1149 | 3 | 145834.0 | 50818 | 2101-10-2310:22:0030 | 30.0 | mmHg | CNUI | 0 |
| 406 | 1163 | a | 145834.0 | 50818 | 2101-10-2316:10:0029 | 29.0 | mmHg | (Null) | 0 |
| 468 | 588 | 3 | 145834.0 | 50818 | 2101-10-2020:04:0028 | 28.0 | mmHg | (Null) | 0 |
| 486 | 606 | 3 | 145834.0 | 50818 | 2101-10-2021:51:0033 | 33.0 | mmHg | (Nuil) | 0 |
| 502 | 622 | 3 | 145834.0 | 50818 | 2101-10-2100:42:0030 | 30.0 | mmHg | (NuII) | 0 |
| 568 | 506 | 3 | 145834.0 | 50818 | 2101-10-2019:12:0040 | 40.0 | mmHg | (Null) | 0 |
| 580 | 518 | 3 | 145834.0 | 50818 | 2101-10-2019:14:0028 | 28.0 | mmHg | (Num | 0 |
| 783 | 189 | 3 | (NuII) | 50818 | 2101-10-1209:18:00338 | 38.0 | mmHg | (Null) | 0 |
| 802 | 208 | 3 | (NuM) | 50818 | 2101-10-1210:51:0039 | 39.0 | mmHg | (Num | 0 |
| 820 | 226 | 2 | (NuM) | 50818 | 2101-10-12 12:05:0044 | 44.0 | mmHg | (NuII) | 0 |
| 836 | 242 | 3 | (NuII) | 50818 | 2101-10-1213:00:0040 | 40.0 | mmHg | (Null) | 0 |
| 853 | 259 | 3 | (Nu 0) | 50818 | 2101-10-1214:22:0042 | 42.0 | mmHg | (NuII) | 0 |
| 1089 | 2509 | 4 | CNUM | 50818 | 2191-05-1813:16:0027 | 27.0 | mmHg | abnormal | 0 |
| 1281 | 687 | 3 | 145834.0 | SO818 | 2101-10-2103:09:0029 | 29.0 | mmHg | (Null | 0 |
| 1313 | 719 | a | 145834.0 | 50818 | 2101-10-2109:46:0026 | 26.0 | mmHg | (Null) | 0 |
| 1322 | 728 | 3 | 145834.0 | 50818 | 2101-10-2109:51:0033 | 33.0 | mmHg | (Null) | 0 |
| 1330 | 736 | 3 | 145834.0 | 50818 | 2101-10-2110:23:0027 | 27.0 | mmHg | (Null) | 0 |
| 1379 | 785 | 3 | 145834.0 | 50818 | 2101-10-2115:48:0028 | 28.0 | mmHg | (NoII) | 0 |
| 1393 | 799 | 3 | 145834.0 | 50818 | 2101-10-2115:52:0036 | 36.0 | mmHg | (Null) | 0 |
| 1409 | 815 | 3 | 145834.0 | 50818 | 2101-10-2117:33:0030 | 30.0 | mmHg | (NuII) | 0 |
| 1424 | 830 | 3 | 145834.0 | 50818 | 2101-10-2117:37:0037 | 37.0 | mmHg | (Nuti) | 0 |
图 1: 初步提取数据图
1.2 数据具体处理
由于 MySQL 时间戳利用的 Unix 时间只能表示到 2038 年, 不便于后续处理, 因此这
段用 Python 语言进行处理。
将上部分的数据导出为 (\mathrm{csv}) 格式,并利用 python 读取。这里首先选择 SUBJECT_ID
为 3 的数据作为示例。
1.3 数据合并
这里首先将 PO2 和 PCO2 中 SUBJECT_ID 相同的数据合并到一个表格中, 并删除其余无关列, 仅保留下时间 CHARTTIME、住院时期 HADM_ID 以及 O2 和 CO2 的值。这里用的数据 SUBJECT_ID 分别为 3 ,
| HADM_ID_X | CHARTTIME | CO2 | 02 | |
|---|---|---|---|---|
| 0 | NaN | 2101-10-12 09:18:00 | 38 | 244 |
| 1 | NaN | 2101-10-12 10:51:00 | 39 | 159 |
| 2 | NaN | 2101-10-12 12:05:00 | 44 | 173 |
| 3 | NaN | 2101-10-12 13:00:00 | 40 | 151 |
| 4 | NaN | 2101-10-12 14:22:00 | 42 | 138 |
| 5 | NaN | 2101-10-12 18:17:00 | 33 | 80 |
| 6 | 145834.0 | 2101-10-20 19:12:00 | 40 | 20 |
| 7 | 145834.0 | 2101-10-20 19:14:00 | 28 | 313 |
| 8 | 145834.0 | 2101-10-2020:04:00 | 28 | 329 |
| 9 | 145834.0 | 2101-10-2021:51:00 | 33 | 287 |
| 10 | 145834.0 | 2101-10-2100:42:00 | 30 | 175 |
| 11 | 145834.0 | 2101-10-21 03:09:00 | 29 | 181 |
图 2: 初步处理数据图
1.4 缺失值处理
可以看到此时的数据中存在一些缺失值, 根据分析, 前部分确实的为一次住院, 最后有
一个数据异常, 因此不妨将缺失的值赋为 1 。
此时准备对结果进行可视化, 发现氧气和二氧化碳的数据值并非为数型, 而是为字符
型, 因此将这两列转为数字型, 此时将数据可视化得到下图:
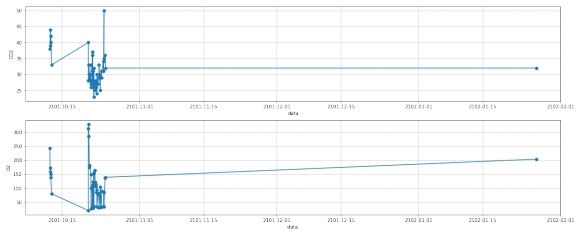
图 3: 离群点处理前
1.5 离群点处理
由于离群点仅有一个, 一个病人再住院期间可以进行一次或者多次血气指标收集, 对于一个病人, 在单次住院期间仅收集一次是无意义的, 也无法进行插值, 只有在一个病人单次住院期间收集多次的情况下, 插值才有意义。因此可将该点去除, 去除后数据如下图所
示。
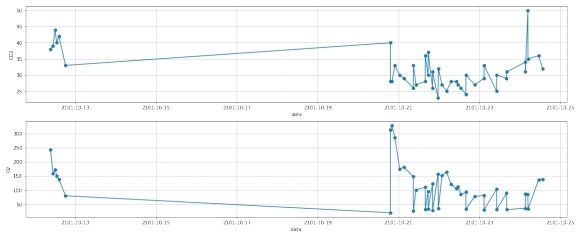
图 4: 离群点处理后
由于数据明显可分为两部分, 因此数据应为两次住院期间的数据, 因此将数据分为两
部分, 即两次住院的数据。
第一部分可视化
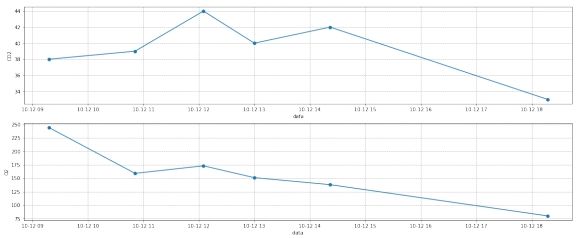
图 5: 一部分去噪前
由于该部分数据量较少, 插值准确率低, 因此不采用第一部分数据进行处理。第二部分可视化的结果如下图所示: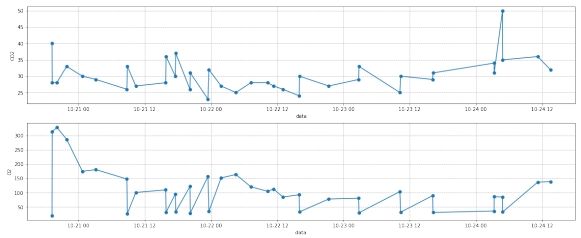
图 6: 二部分去噪前
1.6 去噪
这里使用了 (3\sigma) 原则进行去噪 (\lbrack 1\rbrack) ,由于样本处于 ((\mu {-} 3\sigma,\mu + 3\sigma)) 的概率为 0.9973 。因
此用此原则进行去噪有很大的作用。具体代码如下:
def drop_noisy(df):
(\mathrm{df}) _copy ( = \mathrm{df} {\cdot} \operatorname{copy}())
df_describe = df_copy.describe()
for column in [‘CO2’,’O2’]:
mean ( = ) df_describe.loc [‘mean’,column]
std ( = ) df_describe.loc [‘std’,column]
minvalue ( = ) mean ( {-} 3 * ) std
maxvalue ( = ) mean ( + 3 * ) std
df_copy = df_copy[df_copy[column] >= minvalue]
df_copy = df_copy[df_copy[column] <= maxvalue]
return df_copy
去噪后的结果如下图所示:
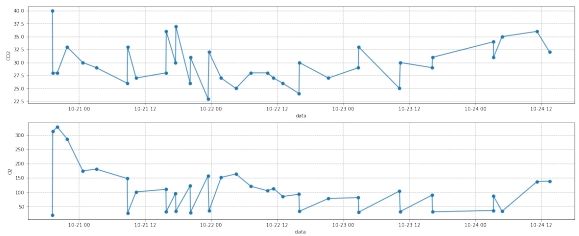
图 7: 二部分去噪后
1.7 插值
在插值地方, 首先将时间一列转为时间戳的形式, 然后分别对每一天的数据进行插值, 这里对于时间使用了线性插值,对 (\mathrm{CO}2) 和 (\mathrm{O}2) 使用了阶梯插值,最后将数据整合到一起, 并将时间戳转化为时间的形式, 从而完成插值。
插值前, 结果如下图所示:
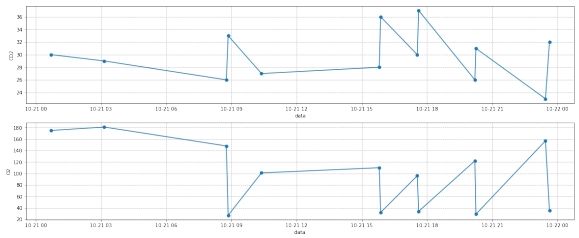
图 8: 样本三第二部分部分数据插值前
经过插值后, 结果如下图所示:
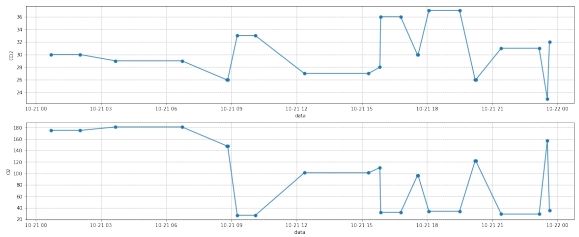
图 9: 样本三第二部分部分数据插值后
2 第二组数据
由上述第一组数据的处理可以得到处理方法, 下述处理同上。
2.1 离群点处理
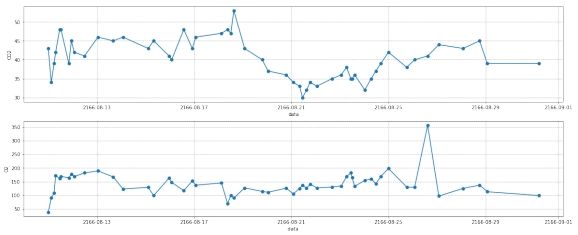
图 10: 第二组数据离群点处理前
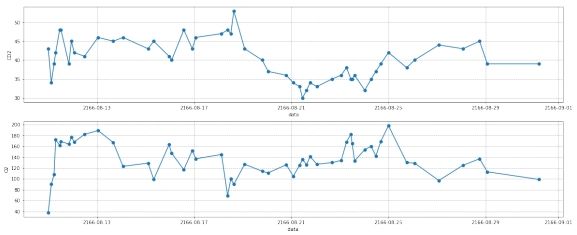
图 11: 第二组数据离群点处理后
2.2 去噪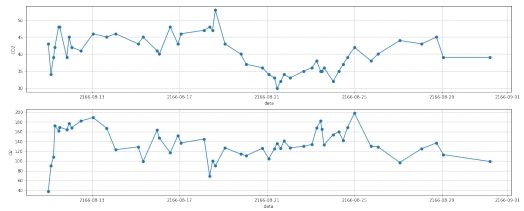
图 12: 第二组数据去噪处理后
2.3 插值
插值前:
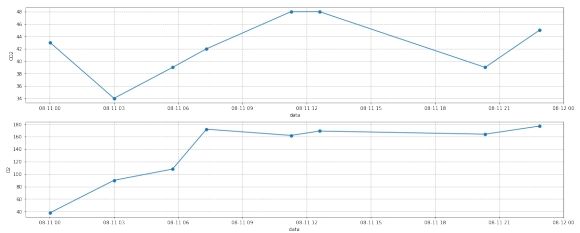
图 13: 第二组数据插值前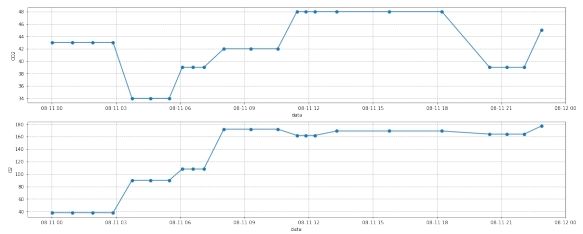
图 14: 第二组数据插值后
3 第三组数据
3.1 离群点处理
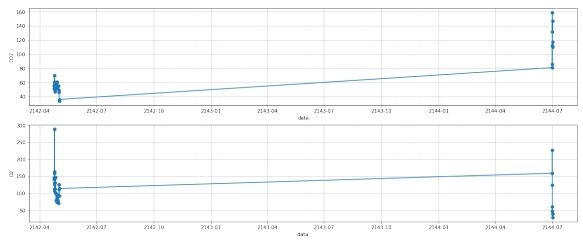
图 15: 第三组数据去离群点前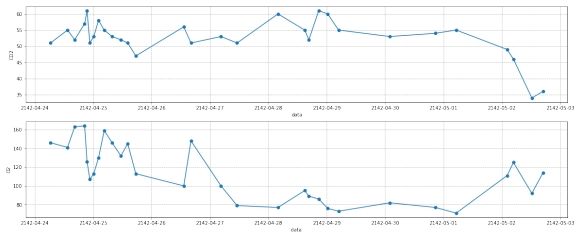
图 16: 第三组数据去离群点后
3.2 去噪
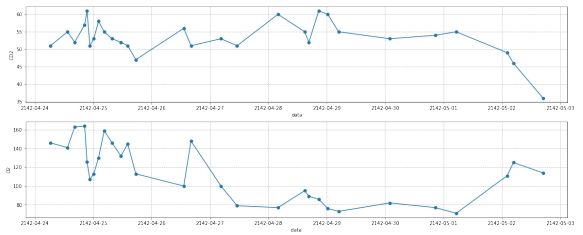
图 17: 第三组数据去噪后
3.3 插值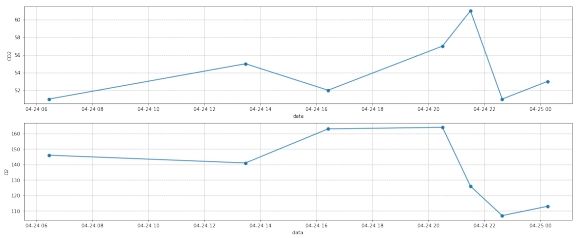
图 18: 第三组数据插值前
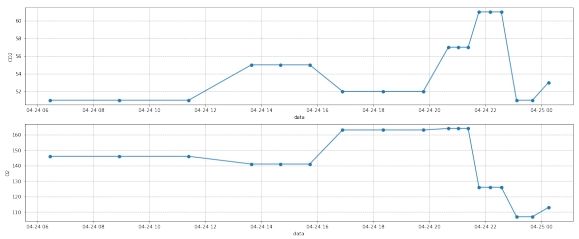
图 19: 第三组数据插值后
4 总结
这次作业在很多地方都有较大的坑, 也是很久以来最难的一次大作业, 以至于做了很久才有一点头绪, 对于我而言, 最难的地方在于插值部分, 由于作为横轴的时间必须经过处理后才能进行相关处理, 在这方面进行插值以及可视化都出现了不小的问题, 经过反复思考并请教相关同学才成功解决。
通过这次数据挖掘的作业, 我感觉自己收获很大, 也学到了很多知识, 我有了一些新的认识, 数据预处理很重要, 未处理过的数据中往往有很多问题, 如果直接拿去用由于数据中存在的很多有问题的地方会使得结果与预期有很大的差异, 因此做好数据预处理是很有必要的一个步骤。在近期的学习中我学习到了数据库以及数据处理方面的很多技巧, 这对于进一步的数据挖掘应该有着不小的帮助, 也有助于今后在该领域的发展。
参考文献
[1] 刘彬, 戴桂平. 基于白化检验和 3 准则的小波阈值去噪算法 [J]. 传感技术学报,2005,18(3):473-476. DOI:10.3969/j.issn.1004-1699.2005.03.008.
A SQL 程序代码 1
提取 PO2 数据
| USE hello_mysql; | SELECT | |
| *: | ||
| FROM | ||
| labevents mini | ||
| WHERE | ||
| T | ITEMID = 490 | |
| M | OR ITEMID = 3785 | |
| 9 | OR ITEMID = 3837 | |
| 0 | OR ITEMID = 50821 |
B SQL 程序代码 2
提取 PCO2 数据
| USE hello_mysql; | SELECT | |
| *: | ||
| FROM | ||
| labevents_mini | ||
| WHERE | ||
| ITEMID = 3784 | ||
| OR ITEMID = 3835 | ||
| OR ITEMID = 50818 |
C 主程序代码
- Data enhancement.py
1 | # -*- coding: utf-8 -*- |
D 作业要求
D. 1 数据集说明
该数据集是一个免费的大型数据库, 包含与 2001 年至 2012 年之某医疗机构重症监护
室收治的 40,000 多名患者相关的健康相关数据。该数据集已进行数据脱敏。
数据集的说明参阅 https://mimic.physionet.org/about/mimic/。此次任务涉及到数据集中的三个表格, PATIENTS, CHARTEVENTS 和 LABEVENTS, 表格的说明分别参考
https://mimic.physionet.org/mimictables/patients/ https://mimic.physionet.org/mimictables/chartevents/ https://mimic.physionet.org/mimictables/labevents/
D. 2 任务说明
本次任务的目的是处理 (\mathrm{pO}2,\mathrm{pCO}2) 两个指标。这两个指标均为病人的血气指标,以一定的时间间隔采集。一个病人一次住院期间可能收集一次或者多次。要求, 按照采集时间的前后顺序,汇总每个病人每次住院期间的所有的 (\mathrm{pO}2,\mathrm{pCO}2) 指标值。涉及到的预处理方法包括插值, 去噪, 缺失值填充, 离群点数据处理, 可视化等。
pO2 和 PCO2 数据存储在 CHARTEVENTS 和 LABEVENTS 两个表格中 (不是分别存储, 而是每个表格都包括这两个指标)。两个表格中以 ITEMID 字段进行标注, 其中 PO2 的 ITEMID 为 (\lbrack 490,3785,3837,50821\rbrack) 之一,PCO2 的 ITEMID 为 (\lbrack 3784,3835,50818\rbrack) 之一。SUBJECT_ID 字段指示不同的病人 (如张三和李四的 SUBJECT_ID 分别为 00001
和 00002), HADM_ID 指示一次住院时期 (一个病人可能多次入院, 同一 SUBJECT_ID, HADM_ID 不同则认为是同一病人不同的住院经历, 在收集数据时需要区分)。





